Ajuda per a GeneticsLab
Aquesta guia us mostrarà com realitzar les tasques més típiques del flux de treball amb GeneticsLab. Seleccionau un tema de la llista per començar. Gaudiu de GeneticsLab!
GeneticsLab us dóna la benvinguda
Aquesta plana de documentació us ajudarà a començar amb el flux de treball típic de GeneticsLab per a iOS. Seleccionau un dels temes de la llista per mostrar la documentació associada.
Recursos d'aprenentatge addicionals
Estic fent feina per a poder afegir documentació addicional en relació al codi genètic i als processos bàsics en l'àmbit de la biologia molecular en el futur. Mentrestant, aquí teniu alguns enllaços a alguns recursos que us poden resultar útils en cas de dubte:
- ADN i ARN a la Vikipèdia
- Processos de transcripció i traducció a la Vikipèdia
- Codi genètic a la Vikipèdia
Aquesta guia també està disponible en altres idiomes:
Tornau a daltL'entorn d'edició
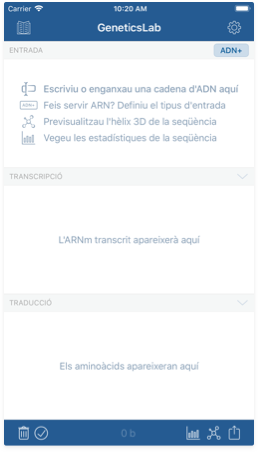
L'àrea de treball és el primer que heu vist en iniciar l'aplicació. Aquí podeu treballar amb les vostres seqüències d'ADN o ARN, que podreu escriure o enganxar en el camp d'entrada. Quan el contingut d'aquest camp canvia, GeneticsLab realitza 4 accions:
- Dóna format automàticament a la vostra seqüència per mostrar-la agrupada en triplets o seguint un altre criteri de la vostra elecció. Consultau 'Preferències generals' per a més informació sobre les opcions disponibles.
- Coloreja els caràcters invàlids en vermell. Aquests són ignorats en el posterior processament. També podreu veure caràcters tenyits de blau que representen caràcters addicionals del codi genètic. Consultau 'Preferències generals' per a més informació sobre aquest tema.
- Actualitza el comptador de bases per reflectir el nombre total de símbols que heu escrit.
- Genera els productes de transcripció i traducció corresponents a la vostra seqüència.
Elements de la interfície
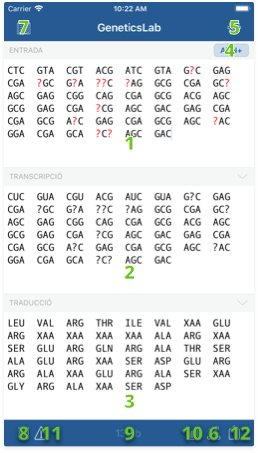
- Camp d'entrada: Aquí és on podeu escriure o enganxar la vostra seqüència.
- Camp de transcripció: Aquí es mostrarà el producte de la transcripció de la vostra seqüència, d'acord amb el tipus d'entrada establert.
- Camp de traducció: Aquí es mostrarà la cadena d'aminoàcids producte de la traducció del contingut del camp de transcripció, utilitzant el codi genètic configurat.
- Indicador del tipus d'entrada: Indica quin tipus d'entrada s'està fent servir per a processar la vostra seqüència. Pitjau-hi per mostrar una llista amb els possibles tipus d'entrada.
- Botó 'Preferències': Feu servir aquest botó per accedir a les preferències de l'aplicació i personalitzar la vostra experiència.
- Botó d'hèlix 3D: Aquest botó inicia la creació d'un model tridimensional de la vostra seqüència.
- Botó 'Referència': Aquest botó us durà a la pantalla de referència.
- Botó 'Esborrar document': Feu servir aquest botó per esborrar el que heu escrit.
- Comptador de símbols: Aquest indicador informa sobre quants nucleòtids hi ha a la vostra seqüència.
- Botó 'Estadístiques': Aquest botó us durà a la pantalla d'estadístiques.
- Botó 'Errades': Aquest botó us durà a la pantalla de revisió d'errades.
- Botó de compartir: Feu servir aquest botó per imprimir o compartir la vostra seqüència per correu electrònic, xarxes socials o altres aplicacions instal·lades al vostre dispositiu iOS.
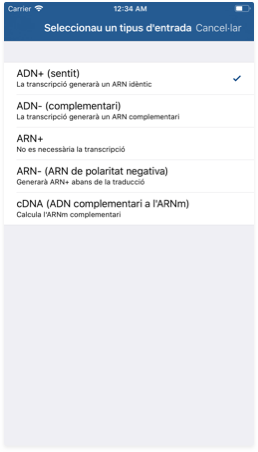
Tipus d'entrada disponibles
- ADN+: Aquest és el mode per defecte. Assumeix que estau introduint la cadena d'ADN amb sentit i, per tant, l'ARN missatger es generarà directament a partir d'aquesta. En aquest mode, els uracils (U) s'interpreten com a timines (T).
- ADN-: Aquest mode assumeix que la vostra seqüència d'entrada és complementària a la cadena amb sentit i, per tant, l'ARN missatger es generarà a partir d'una complementària a la vostra. En aquest mode, els uracils (U) s'interpreten com a timines (T).
- ARN+: Aquest mode assumeix que la vostra seqüència ja és l'ARN missatger que voleu traduïr i, per tant, no hi ha processament addicional. En aquest mode, les timines (T) s'interpreten com a uracils (U).
- ARN-: Aquest mode assumeix que la vostra seqüència és complementària a la seqüència d'ARNm que es traduirà i, per tant, es generarà un ARNm complementari. En aquest mode, les timines (T) s'interpreten com a uracils (U).
- cDNA: Aquest mode assumeix que la vostra seqüència és una cadena d'ADN complementària a l'ARN missatger que es traduïrà. Per tant, es comporta com el mode ADN-.
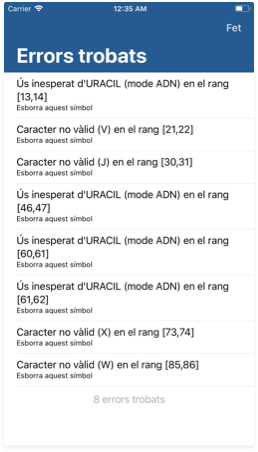
L'inspector d'errors
Detectar i resoldre errors típics a les seqüències d'ADN, ARN o aminoàcids és una de les prestacions més potents de GeneticsLab. Podeu obrir l'inspector pitjant el segon botó del vèrtex inferior dret a la pantalla de l'editor. Actualment, el cercador d'errades pot detectar i resoldre 3 tipus de problemes:
- Caracters invàlids. S'hi inclou qualsevol caracter que no sigui una A, C, G, T, U o qualsevol dels caracters genètics addicionals si el seu ús està habilitat.
- Traducció ambígua. Té lloc quan es processa un codó que no conté la informació suficient per sintetitzar un aminoàcid concret. Teniu en compte que alguns codons poden ser viables tot i no tenir especificada una A, C, G o T a la 3a base, donada la degeneració del codi genètic. Tanmateix, un excés d'inespecificitat pod donar com a resultat una traducció ambígua (no es pot determinar l'aminoàcid).
Correcció automàtica
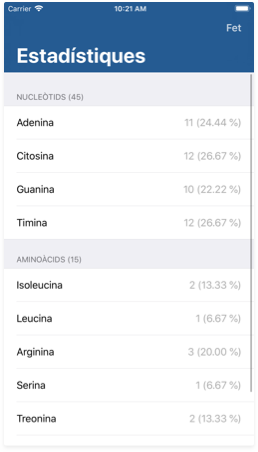
Estadístiques
La pantalla d'estadístiques us permet obtenir informació sobre les proporcions dels diferents símbols a la vostra seqüència d'entrada. Podeu arribar a aquesta pantalla pitjant el tercer botó del vèrtex inferior dret a l'editor.
Cada fila us mostrarà la proporció corresponent al nucleòtid en qüestió, tant en valor absolut com en percentatge.
Tornau a daltPreferències del processament
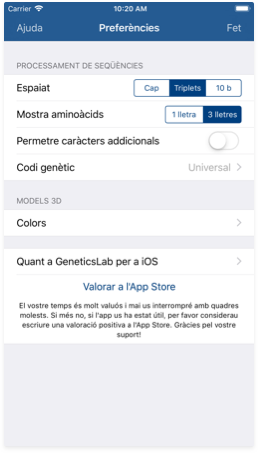
La pantalla de preferències us permet personalitzar la manera en què les vostres seqüències d'entrada són formatejades i processades. Hi podeu accedir pitjant el botó del vèrtex superior dret a l'editor.
Aquests són alguns dels aspectes que podeu personalitzar:
- Espaiat: Feis servir aquest control per personalitzar com s'aplica format a la seqüència d'entrada. Aquí teniu la mateixa seqüència formatejada segons els 3 sistemes:
- Cap
ATGATGATGATGATGATGATGATGATGGGTGATGCTGATCGATCGTAGGATACC - Triplets (per defecte)
ATG ATG ATG ATG ATG ATG ATG ATG ATG GGT GAT GCT GAT CGA TCG TAG GAT ACC - 10 b
ATGATGATGA TGATGATGAT GATGATGGGT GATGCTGATC GATCGTAGGA TACC
- Cap
- Nomenclatura d'aminoàcids: Feis servir aquest control per personalitzar com es mostren els aminoàcids. Aquí teniu la mateixa seqüència formatejada segons els 2 sistemes:
- 1 lletra
MGLVALPQRVTTIYEVSHF|MHGFEGG| - 3 lletres (per defecte)
Met Gly Leu Val Ala Leu Pro Gln Arg Val Thr Thr Ile Tyr Glu Val Ser His Phe END Met His Gly Phe Glu Gly Gly END
- 1 lletra
- Permetre caràcters addicionals: Podeu fer servir aquest control per activar o desactivar el processament de caràcters addicionals en el codi genètic. Podeu trobar més informació sobre aquests caràcters a la taula adjunta. Quan estan deshabilitats, els caràcters addicionals són processats com a caràcters invàlids. Per favor, teniu en compte que fent servir aquests caràcters de forma incorrecta podeu donar lloc a traduccions ambígües.
- Codi genètic: El codi genètic universal hauria de ser suficient per a la majoria de les vostres activitats. Tanmateix, si necessitau fer servir una variant diferent com el codi genètic mitocondrial dels vertebrats, el teniu disponible en aquest menú.
El centre de referència
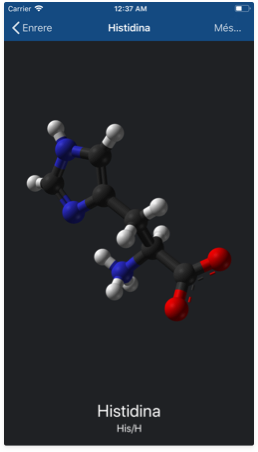
Si sou un principiant en l'àmbit de la genètica, felicitats! Acabau de descobrir un món increïble, però potser al principi no recordeu alguna cosa sobre un aminoàcid concret o sobre un nucleòtid. El centre de referència us ajudarà com a eina ràpida de consulta en aquest cas. Hi podeu accedir pitjant el botó 'Referència' (el 2n començant per l'esquerra a la barra inferior) des de l'àrea de treball.
Accedireu a una llista amb els 20 aminoàcids que conformen el codi genètic universal. També podreu accedir a la llista de nucleòtids pitjant la secció corresponent del control que trobareu a la part superior. Des d'aquí, si sel·leccionau qualsevol molècula, accedireu a una pàgina amb detalls on hi trobareu:
- Una imatge de la molècula en qüestió.
- El nom complet de la molècula, així com les abreviacions estandaritzades que es fan servir per a referir-s'hi.
- El botó 'Més…'. Pitjau-lo per accedir a l'article corresponent a la molècula que heu sel·leccionat a la Viquipèdia.
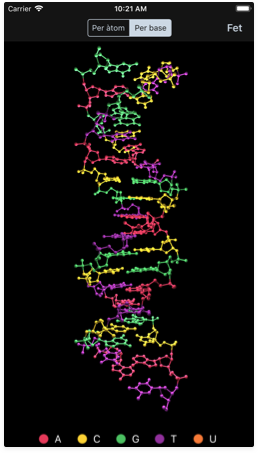
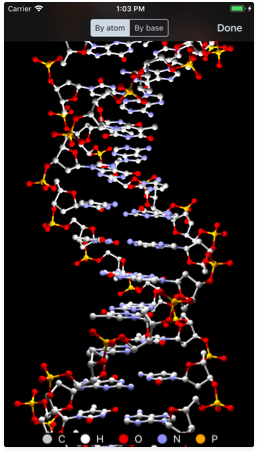
Generant models 3D
Aprendre visualment és encara millor i ara podeu experimentar amb magnífics models 3D de les vostres seqüències. Feis tap en el botó de la molècula al cantó inferior dret i GeneticsLab construirà un model 3D de l'estructura helicoïdal de la vostra seqüència, sia d'ADN o d'ARN.
Un cop generada l'estructura d'hèlix veureu com comença a girar automàticament. Per aturar l'animació, tocau qualsevol punt de la pantalla.
Experimentau
No es tracta només de generar una hèlix 3D de la vostra seqüència. Podeu moure-la, rotar-la i ampliar-la per descobrir cada detall. Aquests són els gestos que teniu disponibles:
- Pessigau per apropar o allunyar la molècula.
- Feis un gir amb dos dits per rotar la molècula.
- Lliscau amb un dit per moure la càmera al voltant de la molècula.
- Lliscau amb dos dits per moure la molècula al llarg de la pantalla.
També teniu disponibles diferents esquemes de color:
- Per àtom: acoloreix cada àtom d'acord amb el seu element de la taula periòdica.
- Per base: acoloreix cada àtom d'acord amb la base nitrogenada a la qual pertany.
Personalitzau
Podeu escollir el sistema de color per defecte o fins i tot els colors individuals a la pantalla de Preferències. Vegeu 'Preferències 3D' per conèixer més detalls.
Tornau a daltPreferències de color en models 3D
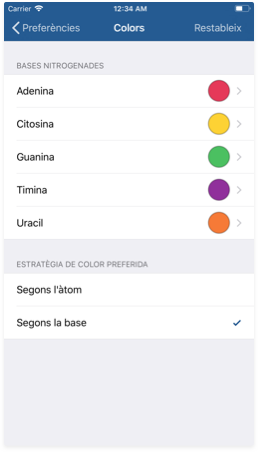
La pantalla de preferències us permet personalitzar la forma en què es generen els models 3D. Podeu accedir a ella des de l'editor fent tap en el botó del cantó superior dret. Després accediu a la secció corresponent fent servir el botó 'Colors'.
Colors per defecte
Aquests són els valors per defecte per al sistema de color 'Per àtom'. Actualment no poden modificar-se a la pantalla de preferències.
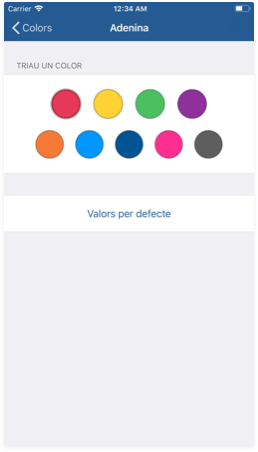
Per al sistema 'Per base' podeu modificar el color de cada base nitrogenada fent servir el selector de color. Seleccionau els colors que volgueu i provau-los al visor 3D.
Feis servir el botó 'Restableix' per restaurar tots els colors als seus valors per defecte, que són els següents:
Colors per defecte
Podeu escollir el sistema de colors inicial per a la generació de models 3D en aquesta secció. Aquesta configuració només afecta al pintat inicial que s'aplica en obrir el visor 3D, podreu seguir canviant entre ambdós sistemes en qualsevol moment.
Tornau a daltTeniu quelcom a dir?
Si teniu qualque pregunta, queixa o suggeriment, per favor no dubteu en posar-vos en contacte amb jo.